TWOJA PRZEGLĄDARKA JEST NIEAKTUALNA.
Wykryliśmy, że używasz nieaktualnej przeglądarki, przez co nasz serwis może dla Ciebie działać niepoprawnie. Zalecamy aktualizację lub przejście na inną przeglądarkę.
Data: 27.01.2023 Kategoria: medycyna, nauka/badania/innowacje, Wydział Podstawowych Problemów Techniki

Białka są jednym z najważniejszym składników naszego organizmu a ich właściwości i wpływ na zdrowie wciąż są badane. Naukowcy z Katedry Inżynierii Biomedycznej na Wydziale Podstawowych Problemów Techniki sprawdzają, jak przewidzieć, które z nich mają tendencje do tworzenia tzw. agregatów amyloidowych i jaki wpływ mają na to ich interakcje z innymi białkami o podobnych właściwościach.
Nawet 20% masy ciała człowieka stanowią białka. Badania pokazują, że wiele chorób neurodegeneracyjnych, uważanych za choroby cywilizacyjne, ma związek z łączeniem się białek lub ich fragmentów w długie włókna. Białka, które biorą udział w ich tworzeniu nazywane są amyloidami. Większość przypadków amyloidów to efekt błędnego zwinięcia łańcucha białka, na przykład wówczas, gdy nastąpi częściowe rozwinięcie łańcucha białkowego lub podzielenie go na fragmenty, które prowadzi do odsłonięcia obszarów o skłonnościach amyloidowych.
– Niestety, te struktury są niesłychanie trudno rozpuszczalne i mogą być szkodliwe dla komórek, szczególnie w bardzo wczesnej fazie tworzenia włókna - wyjaśnia prof. Małgorzata Kotulska z Wydziału Podstawowych Problemów Techniki. – Istnieją jednak też funkcjonalne białka amyloidowe, na przykład bakteryjne, których funkcja wymaga właśnie takiej struktury fibrylarnej i jest ona dla nich naturalna. U ludzi takie funkcjonalne amyloidy to jednak rzadkość.
– Takie struktury znaleziono w mózgu chorych na chorobę Alzheimera, jak również przy chorobie Parkinsona, stwardnieniu zanikowym bocznym, chorobie Huntingtona, zaćmie, a nawet w cukrzycy typu 2 i wielu innych chorobach – wylicza prof. Małgorzata Kotulska.
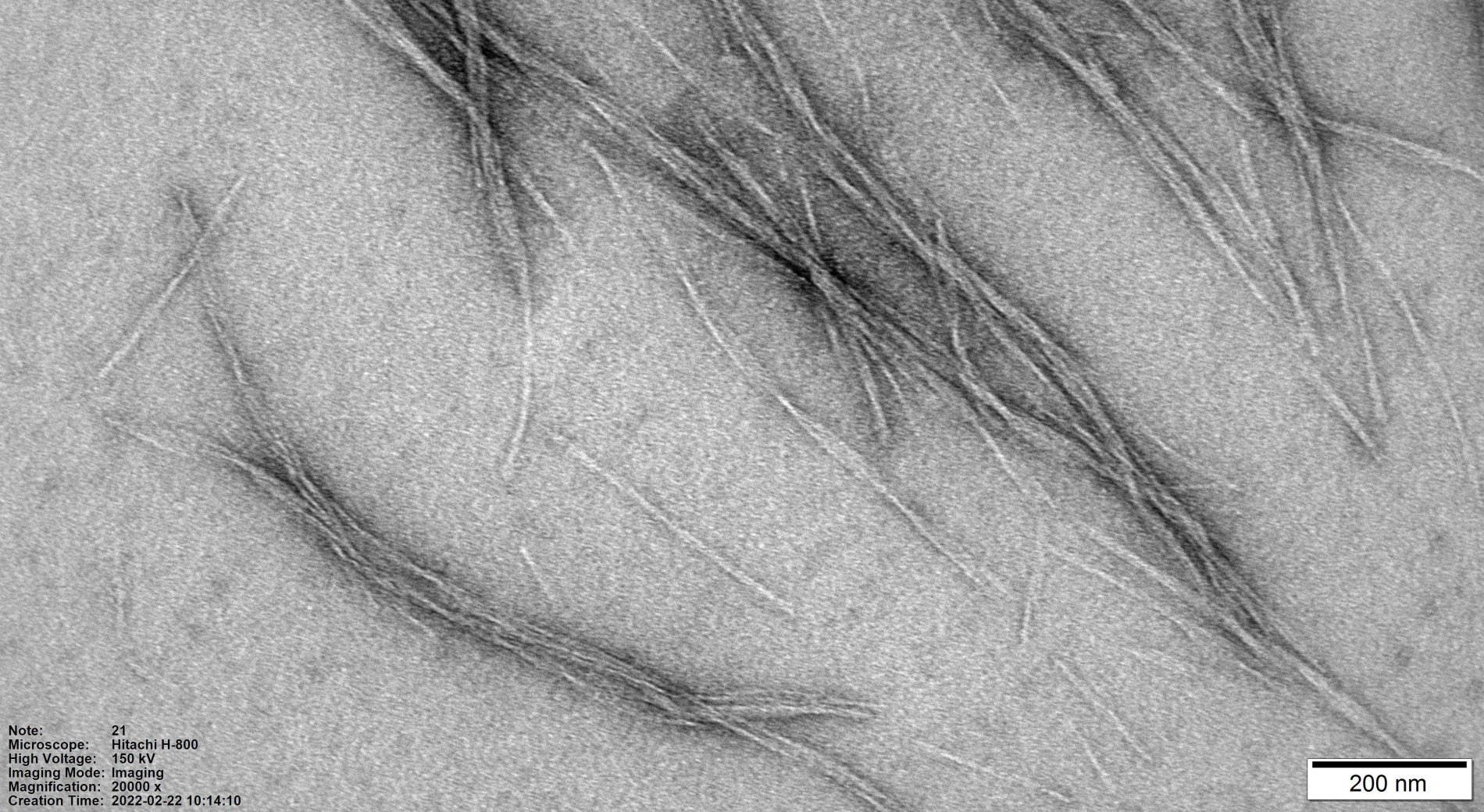
fot. Włókno amyloidowe pod mikroskopem elektronowym (autor dr Andrzej Żak)
Ale to nie wszystko. Niektóre amyloidy mogą wpływać na agregację innych białek. Może to nastąpić w wyniku interakcji pomiędzy łańcuchami o różnym składzie aminokwasowym.
– Zjawisko to nie zostało dotychczas dobrze poznane – wyjaśnia prof. Kotulska. – Na ogół obserwuje się je przy pewnym podobieństwie w sekwencji aminokwasów w oddziałujących ze sobą łańcuchach białkowych. Jednak nie zawsze ten warunek jest konieczny.
Do dziś badacze na całym świecie zastanawiają się, czy takie interakcje pomiędzy białkami są częste i mogą prowadzić do procesów chorobowych. Dzięki projektowi „Wyznaczenie wzorów interakcji krzyżowych między białkami amyloidowymi”, którym kieruje prof. Małgorzata Kotulska z Katedry Inżynierii Biomedycznej, być może poznamy odpowiedź na te pytanie. Jego realizację finansuje Narodowe Centrum Nauki w ramach grantu Opus.
Prof. Małgorzata Kotulska badania nad amyloidami rozpoczęła już w 2010 roku, odbywając staż na Yale University w New Haven, finansowany przez stypendium fundacji Fullbrighta. – Zajmowałam się tam modelowaniem struktury białek i analizą miejsc kontaktowych w kanałach białkowych – wyjaśnia badaczka z W11. – Tam też natrafiłam na artykuł, w którym wyjaśniano, jak za pomocą charakterystyki kontaktów między aminokwasami w białku, można odkrywać tendencje amyloidowe. To właśnie wtedy – także z przyczyn osobistych, gdyż w rodzinie wystąpiła choroba neurodegeneracyjna – ten temat mnie wciągnął – mówi prof. Kotulska.
 Po powrocie na PWr, razem ze swoimi doktorantami i kolegami, zaczęła dokładnie badać tematy związane z modelowaniem białek amyloidowych.
Po powrocie na PWr, razem ze swoimi doktorantami i kolegami, zaczęła dokładnie badać tematy związane z modelowaniem białek amyloidowych.
Prof. Kotulska: – To była czysto obliczeniowa praca, ale zaczęły nam wychodzić ciekawe rzeczy. Dostałam wtedy grant na kontynuację badań związanych z miejscami kontaktowymi, w którym niepostrzeżenie coraz więcej miejsca zaczęły zajmować badania nad przewidywaniem białek amyloidowych. Stworzyliśmy pierwsze nasze narzędzia do badania amyloidów, takie jak: Fish Amyloid – do przewidywania, czy białka mają tendencje do tworzenia agregatów amyloidowych i AmyLoad – baza danych wszystkich znanych białek i peptydów amyloidowych, które udostępniliśmy w Internecie.
Temat przyciągnął współpracowników zewnętrznych. Wspólnie z dr. Michałem Burdukiewiczem (wówczas Uniwersytet Wrocławski, obecnie Autonomous University of Barcelona i Uniwersytet Medyczny w Białymstoku) nasi naukowcy opracowali kolejne narzędzie do przewidywania własności amyloidowych białek, AmyloGram. Metoda ta wykazuje bardzo wysoką skuteczność działania, jednak nadal ograniczoną głównie do amyloidów patologicznych, których znamy nieporównanie więcej niż amyloidów funkcjonalnych.
Badacze stwierdzili, że badania warto rozwinąć też w stronę eksperymentalną. Wsparcie, które otrzymali w postaci grantu od Narodowego Centrum Nauki pozwala im sprawdzać możliwości przewidywania interakcji między różnymi białkami i peptydami o właściwościach amyloidogennych oraz budować metody obliczeniowe do przewidywania ich interakcji.
Według niektórych teorii, część białek o właściwościach amyloidogennych, które są wytwarzane przez mikrobiom (ogół bakterii znajdujących się w ludzkim jelicie), może się łączyć z ludzkimi białkami wykazującymi pewne tendencje do tworzenia agregatów i tę agregację przyspieszać.
Jednym z czynników, który miałby np. wywoływać chorobę Parkinsona, jest agregacja białka o nazwie alfa-synukleina. – Istnieje teoria, która zakłada, że agregacja ta może być przyspieszona przez białka biofilmowe, przypominające amyloidy znane z ludzkiego organizmu, ale naturalne dla bakterii, które żyją w naszym mikrobiomie – mówi prof. Kotulska.
 Także nasza wiedza o ludzkim mikrobiomie wciąż jest niewystarczająca. Jakie gatunki bakterii, w jakich sytuacjach są dominujące, co dokładnie wytwarzają, które produkty ich metabolizmu są dla nas przydatne, a które niekorzystne – na te pytania również nie ma dokładnych odpowiedzi.
Także nasza wiedza o ludzkim mikrobiomie wciąż jest niewystarczająca. Jakie gatunki bakterii, w jakich sytuacjach są dominujące, co dokładnie wytwarzają, które produkty ich metabolizmu są dla nas przydatne, a które niekorzystne – na te pytania również nie ma dokładnych odpowiedzi.
– To co wiemy na pewno to fakt, że bakterie, z którymi współistniejemy, produkują bardzo dużo swoich metabolitów, które nieraz mocno na nas mocno oddziałują. Można powiedzieć, że nosimy w sobie taką trochę fabrykę leków albo trucizn dla naszego organizmu – wyjaśnia prof. Małgorzata Kotulska.
Dlatego tak ważna wg naszych naukowców byłaby wiedza o tym, jaki typ białek może oddziaływać negatywnie na ludzkie białka agregujące. W połączeniu z tym, co już wiemy o naszych organizmach, mogłoby to pozwolić pomyśleć o usuwaniu tych szkodliwych gatunków bakterii.
Zespół prof. Kotulskiej postawił więc przed sobą szereg pytań, na które teraz chce znaleźć odpowiedzi. Kiedy takie interakcje mogą zaistnieć? Jakie właściwości powinny mieć te białka? Jak wyglądają takie agregaty? Czy one są częste? Czy rzeczywiście między nimi dochodzi do interakcji?
Aby odpowiedzieć chociaż na część z nich badacze z PWr, wspólnie z lekarzami i dr. Burdukiewiczem, stworzyli unikalną w skali światowej bazę danych wszystkich dobrze udokumentowanych eksperymentów dotyczących interakcji amyloidów. Baza AmyloGraph jest w pełni darmowa i dostępna online. Artykuł o powstaniu tej bazy pojawił się w renomowanym czasopiśmie Nucleic Acids Research.
– Stworzyliśmy ją po to, aby sprawdzić czego możemy się spodziewać na podstawie eksperymentów, które już ktoś kiedyś przeprowadził. Mieliśmy nadzieję, że będziemy tu mogli zastosować metody uczenia maszynowego. Ale w tej chwili analizy wskazują, że inne metody mogą się okazać lepsze – wyjaśnia prof. Kotulska.
 Okazało się, że takich danych jest co prawda dużo, ale dotyczą tylko tych białek, które badaczy najbardziej interesowały ze względu na ich udział w chorobach. Więc nie dają one pełnej informacji o właściwościach fizyko-chemicznych tych interakcji. Dlatego naukowcy postanowili podejść do tych badań od strony modelowania strukturalnego. I w efekcie stworzyli zupełnie nowe narzędzie: PACT (Prediction of Amyloid Cross-Interaction by Threading).
Okazało się, że takich danych jest co prawda dużo, ale dotyczą tylko tych białek, które badaczy najbardziej interesowały ze względu na ich udział w chorobach. Więc nie dają one pełnej informacji o właściwościach fizyko-chemicznych tych interakcji. Dlatego naukowcy postanowili podejść do tych badań od strony modelowania strukturalnego. I w efekcie stworzyli zupełnie nowe narzędzie: PACT (Prediction of Amyloid Cross-Interaction by Threading).
- Posiadane już przez nas dane dotyczyły białek, których forma amyloidowa jest efektem przyjęcia nieprawidłowej struktury – mówi prof. Kotulska. – Ale interesują nas także białka, które mają właściwości amyloidowe naturalnie. To jest ich normalna funkcja, one są ewolucyjnie przygotowane na to, żeby przyjmować takie struktury włókniste. Nie zawsze można je przewidywać identycznymi metodami jak amyloidy patologiczne.
Obecnie we współpracy z dr Moniką Szefczyk z zespołu prof. Łukasza Berlickiego z Wydziału Chemicznego syntezowane są krótkie białka i peptydy, na których zespół prof. Kotulskiej sprawdza wyniki przewidywania modeli obliczeniowych. W politechnicznych laboratoriach badane jest m.in. to, czy przez obecność pewnych białek lub peptydów, mogących się pojawić w organizmie ludzkim, inne białko agreguje łatwiej.
– W naszym modelu widzimy, kiedy dwa białka powinny wchodzić w interakcje wzajemnie wpływając swoje skłonności do agregacji, ale wciąż nie mamy wiedzy, czy to spowoduje przyspieszenie czy spowolnienie tworzenia włókien – wyjaśnia badaczka z W11.
Projekt „Wyznaczenie wzorów interakcji krzyżowych między białkami amyloidowymi” wciąż trwa. Teraz badania są na etapie eksperymentalnej weryfikacji modelu zbudowanego w oparciu o dane zgromadzone w bazie AmyloGraph. Pełne rezultaty poznamy nie wcześniej niż we wrześniu 2024 r.
A co zostanie uznane za sukces? – Gdyby się okazało, że nasze narzędzia do przewidywania takich interakcji są skuteczne, potwierdzalne eksperymentalnie, a do tego udałoby się nam wytypować i potwierdzić określoną liczbę nieznanych interakcji między białkami ludzkimi a biofilmowymi bakteryjnymi oraz mielibyśmy znaleźli jasne i klarowne przesłanki, jak ten mechanizm działa – wtedy będę usatysfakcjonowana – mówi prof. Kotulska.
***
Zespół prof. Małgorzaty Kotulskiej, działający w Katedrze Inżynierii Biomedycznej na WPPT, powstał w 2008 roku. Zajmuje się problemami biologii molekularnej w zastosowaniach diagnostyki i terapii medycznej. Dotychczasowa tematyka obejmowała modelowanie bioinformatyczne białek, metody elektrochemioterapii nowotworów, a aktualnie rozpoznawanie amyloidów i ich interakcje.

Obecnie w zespole działa 7 osób, oprócz kierowniczki grupy są to: dr inż. Marlena Gąsior-Głogowska (eksperyment), dr Aleksandra Kalitnik (eksperyment), mgr inż. Alicja Nowakowska (modelowanie), mgr inż. Oliwia Polańska (eksperyment), mgr inż. Natalia Szulc (eksperyment) i mgr inż. Jakub Wojciechowski (modelowanie).
O działalności grupy można też na bieżąco czytać na profilu KotulskaLab na Facebooku.
Autorem wszystkich zdjęć jest dr Mateusz Popek.
Nasze strony internetowe i oparte na nich usługi używają informacji zapisanych w plikach cookies. Korzystając z serwisu wyrażasz zgodę na używanie plików cookies zgodnie z aktualnymi ustawieniami przeglądarki, które możesz zmienić w dowolnej chwili. Ochrona danych osobowych »